JALUR LINTAS GENETIK PURBA
ANTARA ASIA DAN PASIFIK
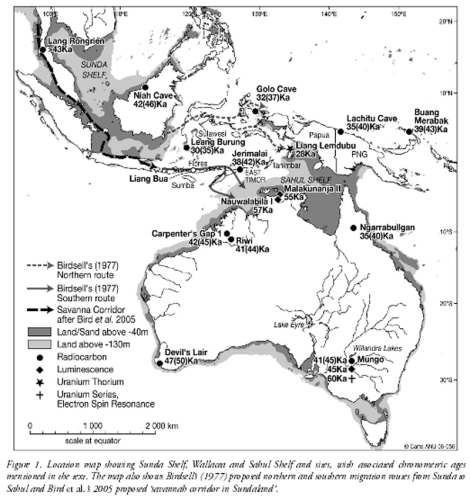
Keragaman genetik, etnis dan bahasa yang tinggi di Kepulauan Nusantara tak terbantahkan lagi. Kehadiran manusia modern di kepulauan Nusantara paska letusan Toba sekitar 74.000 tahun yang lalu bisa ditelusuri dari jejak-jejak penanda genetik pada populasi Indonesia saat ini, dari Sabang di ujung barat sampai Merauke di ujung timur, serta dari kepulauan Sangihe-Talaud di utara sampai pulau Rote di selatan. Namun sangat kurangnya penelitian genetik populasi Indonesia, walau ada beberapa penelitian yang dilakukan secara parsial, membuat rekonstruksi sejarah populasi kepulauan Nusantara agak rumit. Namun dari data-data parsial tersebut, setidaknya bisa dibuat gambaran besar sejarah populasi Indonesia sejak paska letusan Toba sampai masa sejarah.
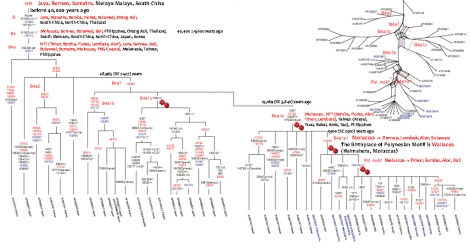
Secara umum, sejarah populasi Indonesia dibagi dalam lima gelombang migrasi yang diawali dengan migrasi populasi Negrito (populasi yang memiliki karakterphenotype seperti Orang Asli di semenanjung Melayu), kemudian setelah sekian lama menghuni Sundaland, sebagian populasi Negrito tersebut mulai meninggalkan Sundaland menuju Sahul (yang kemudian sering disebut dengan populasi Papua/Melanesia/Australoid) dan ke arah utara menuju daratan Asia Tenggara (yang selanjutnya menjadi leluhur populasi penutur Austroasiatik dan Austronesia). Kemudian ketika Last Glacial Maximum (LGM) terjadi, sebagian dari populasi penutur Austroasiatik, yang menghuni daratan Asia Tenggara, kembali migrasi ke kepulauan Nusantara. Dan pada pertengahan era Holocene, penutur Austronesia juga kembali ke kepulauan Nusantara dan menyebar ke arah Pasifik. Gelombang migrasi terakhir terjadi pada masa sejarah, dengan hadirnya pendatang-pendatang dari Arab, Persia, Gujarat, India selatan dan timur, yang meninggalkan jejak genetik cukup kuat pada populasi Nusantara saat ini.
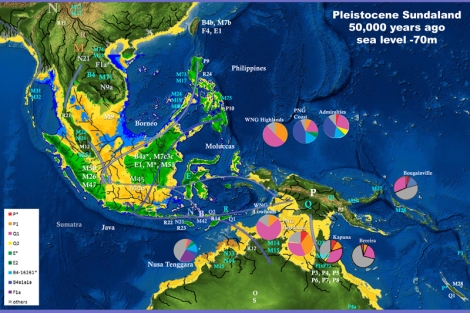
Keragaman populasi Indonesia dipengaruhi oleh perubahan iklim yang ekstrim di era Pleistocene. Populasi pribumi Sundaland harus berhadapan dengan perubahan geografi yang sangat dinamis ketika itu. Mereka meninggalkan Sundaland, walaupun kemudian kembali lagi setelah ribuan tahun, ketika permukaan air laut saat itu sekitar 70 meter di bawah permukaan laut (d.p.l.) saat ini, kemudian turun lagi menjadi 120 meter d.p.l., dan kembali naik ketika es di kutub utara mulai mencair ~17-14.000 tahun yang lalu, bisa menjadi faktor utama terjadinya migrasi; dan ketika puncaknya ~4200 tahun yang lalu, permukaan air laut naik sampai 2 meter d.p.l. saat ini, ketika itulah penutur Austronesia mulai menyebar ke seluruh penjuru Indonesia, kemudian ke Polynesia di Pasifik sampai Madagascar di Afrika. Perubahan tradisi dari pemburu menjadi petani bisa jadi bukanlah faktor utama migrasi, karena mereka terpaksa meninggalkan daerah asalnya karena saat itu bisa jadi mereka mendiami pesisir pulau yang mulai tenggelam karena naiknya permukaan air laut. Jika mereka mendiami dataran tinggi atau pegunungan, mereka tidak akan terdampak oleh mencairnya es di pertengahan Holocene. Hal ini dibuktikan oleh populasi-populasi yang terisolasi seperti di pegunungan Papua, Nias, Mentawai, Sumba, pedalaman Borneo, Nusa Tenggara, yang secara genetik memiliki penanda yang tidak tercampur oleh para petani pendatang (bukti adanya pergeseran genetik, dan populasi yang terisolasi).
Bagaimana mungkin bangsa dengan genetik, etnis dan bahasa yang begitu beragam tidak banyak dilakukan penelitian sejarah populasi dengan multi-disiplin keilmuan?
Penelitian sebelumnya sedikit membantu kita memahami bagaimana struktur populasi di Indonesia (Cox, M.P. , 2005: Indonesian mitochondrial DNA and its opposition to a Pleistocene era origin of proto-Polynesians in Island Southeast Asia; Cox, M.P., 2008: The genetic environment of Melanesia: Clines, clusters and contact; Cox & Lahr, 2006: Y-Chromosome diversity is inversely associated with language affiliation in paired Austronesian and Papuan-speaking communities from Solomon Islands; Cox et al. (2007): A Polynesian motif on the Y-chromosome: population structure in Remote Oceania; Karafet et al. (2010): Major east-west division underlies Y-chromosome stratification across Indonesia; Karafet et al. (2005): Balinese Y-chromosome perspective on the peopling of Indonesia: genetic contributions from pre-Neolithic hunter-gatherers, Austronesian farmers, and Indian traders; Kayser,M (2010): The human genetic history of Oceania: near and remote views of dispersal; Kayser et al. (2006): Melanesian and Asian origins of Polynesians: mtDNA and Y-chromosome gradients across the Pacific; Kayser et al. (2003): Reduced Y-chromosome, but not mitochondrial DNA, diversity in human populations from West New Guinea). Beberapa ilmuwan khusus meneliti sejarah populasi melalui penanda genetik maternal (mtDNA), seperti Hill et al. (2007): A mitochondrial stratigraphy for island southeast Asia; dan Soares et al. (2008): Climate change and postglacial human dispersals in Southeast Asia. Sedangkan penanda genetik paternal (Y-chromosome) cukup rumit dalam mengungkap sejarah populasi Indonesia (Karafet et al., 2014: Refined structure in haplogroup K–M526). Dan dalam penelitian di wilayah Asia Tenggara, populasi Indonesia selalu menjadi sample, dan informasi dari penanda genetik paternal bisa kita dapatkan. Walaupun penanda autosomal cukup berhasil mengubah cara pandang kita terhadap prasejarah suatu wilayah tertentu (yang dengan mudah mendeteksi adanya percampuran antara dua populasi yang berbeda), namun kedua penanda genetik tersebut bisa menjadi alat yang sangat berguna untuk memahami sejarah populasi masa lampau.
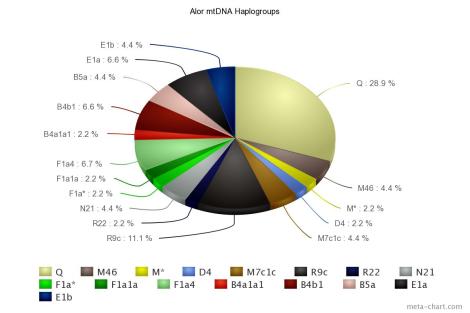
Kemudian, Tumonggor et al. (2013) dalam penelitiannya berhasil mengungkap informasi penting terkait dengan keragaman penanda genetik maternal populasi Indonesia, yang berkisar 0.862-0.996, yang berarti bahwa sebagian besar populasi Indonesia mewarisi garis keturunan maternal (mtDNA) yang unik. Populasi paling beragam ditemukan di Indonesia tengah (Wallacea) seperti Sumba, Flores, Pantar dan Alor, dimana kedua garis keturunan Asia dan Papua hidup berdampingan. Keragaman populasi di Nias dan Mentawai adalah yang paling rendah, serendah kelompok etnis Taiwan (0,838-0,924), bahkan jika dibandingkan dengan populasi garis keturunan Asia di sekitarnya. Artinya, kedua populasi tersebut terisolasi sejak kedatangan mereka di pulau tersebut. Percampuran dengan populasi dari daerah lain sangat minim terjadi di Nias dan Mentawai. Hal yang sama juga terjadi pada populasi pribumi Taiwan. Analisa mtDNA mengungkap pertumbuhan populasi cukup tinggi terjadi pada populasi Bali, Sulawesi, Sumba dan Flores. Sedangkan di Indonesia bagian barat dan timur tidak terlalu mencolok. Setidaknya ada 51 mtDNA haplogroup teridentifikasi, dengan garis keturunan macrohaplogroup M sebesar 47,05% dan macrohaplogroup N sebesar 52,95%.
Frekuensi haplogroup terdeteksi berbeda antara Indonesia bagian barat dan timur. Di bagian barat, mtDNA haplogroup B5a (12%), B4c1b3 (9%), dan Y (10,5%) diwarisi sepertiga populasi. Haplogroup tersebut banyak ditemui di Indonesia bagian barat namun sangat jarang di bagian timur. Di bagian timur, mtDNA haplogroup F1a4 (8,7%), Q termasuk Q1 dan Q2 (7,7%), P (2,8%) dan B4a1a1a (2,3%) mewakili hampir seperempat populasi. Haplogroup tersebut jarang bahkan absen, di bagian barat, karena memang merupakan garis keturunan yang berasal dari Papua (autochthonous, P dan Q), namun kehadiran mtDNA B4a1a1a (Polynesian motif) cukup menarik. Polynesian motif (PM) ditemukan di bagian barat hanya sejauh Bali, meskipun bisa jadi PM sempat mampir di Jawa, Sumatra, Borneo, Nias, dan Mentawai, ketika mereka menuju Madagascar.
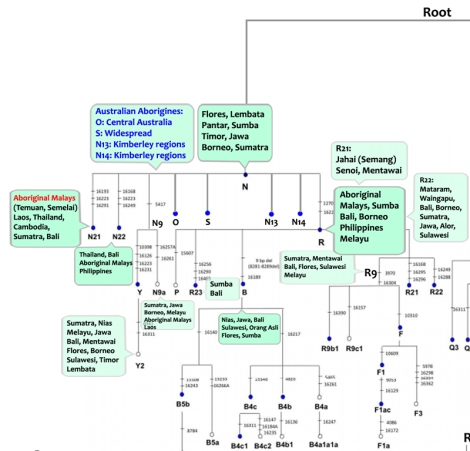
Jika dibandingkan, sebaran mtDNA haplogroup di Indonesia dengan wilayah sekitarnya, sebagian besar memiliki haplogroup yang sama. Garis keturunan maternal yang sangat tua seperti M17a, M73, M47, N21, N22, R21, R22 dan R23tersebar di daratan Asia Tenggara, Filipina dan kepulauan Nusantara, mencerminkan bahwa mereka adalah garis keturunan maternal dari penghuni awal prasejarah Sundaland. Empat dari haplogroup tersebut (M17a, N21, R22, dan R23) ditemukan dengan frekuensi lebih tinggi di bagian barat dibandingkan bagian timur.
Sebelumnya, haplogroup M17, M21, M22, M51, M71, M72, M73, N21, R22, and R23 yang ditemukan di Asia Tenggara dianggap sebagai komponen garis keturunan maternal yang sangat tua di wilayah Asia Tenggara (Macaulay et al. 2005; Hill et al. 2006, 2007; Tabbada et al. 2010). Peng et al. (2010) menguji haplogroup tersebut menggunakan analisa mitogenome dan mengkonfirmasi bahwa M17, M21, M22, M50, M51, M72, M73, M77, N21, R22, and R23 memang haplogroup eksklusif, hanya ditemukan di Asia Tenggara dan bisa ditelusuri asal-usulnya di sekitar Sundaland.
Mari kita telusuri mtDNA haplogroup yang sangat tua tersebut.
M17a (dari garis keturunan M17, sebelumnya dikenal dengan M45. M17 hanya satu mutasi dari macro-haplogroup M pada np 16209, dan M17a hanya satu mutasi dari M17. Artinya M17 muncul minimal 8000-10.000 tahun setelah mtDNA M mendiami Sundaland) ditemukan di Nias, Mentawai, Jawa, Bali, Lombok, Sumba, Flores, Alor, Pantar, Sulawesi dan Maluku, di Mentawai dengan frekuensi tertinggi (12,7%). M17a juga di Filipina (Luzon) dan Semenanjung Melayu.
M73 ditemukan di Bali, Sumba, Flores, Pantar dan Sulawesi. Dengan frekuensi lebih tinggi berada di Wallacea. M73 dan M79 adalah garis keturunan dari M73’79, dengan sebaran M73 di Filipina (dalam bentuk M73a di populasi Visayas), juga M73a di Timor Leste , Vietnam dan juga Laos, sedangkan M79 di China Selatan. Meskipun M73 tersebar luas, terdapat bukti garis keturunan yang beragam dalam garis keturunannya, mencerminkan adanya evolusi regional dari mtDNA M73. M73 hanya satu mutasi dari macro-haplogroup M pada np 16278.
M47 (lima mutasi dari macro-haplogroup M) hanya ditemukan di Sumba dengan frekuensi sangat rendah. Mengapa hanya ada di Sumba? Apakah mereka garis keturunan yang hampir punah? Ini yang disebut genetik terisolasi. Mereka hidup dalam kelompok kecil, menjaga keragaman genetik tetap tinggi dengan tidak kawin dengan populasi lain. Mereka bisa jadi mutasi dari populasi mtDNA M yang migrasi menuju Sahul, namun memisahkan diri ketika mereka sampai di sekitar Nusa Tenggara. M47, bersama M45 dan M46, tidak ditemukan di tempat lain di dunia. mtDNA tersebut berumur sangat tua, bisa jadi lebih dari 50.000 tahun, tergantung umur macro-haplogroup M sebagai referensi.
N21 (garis keturunan tertua macrohaplogroup N, hanya berselang 3 mutasi), kemudian disusul O, N22 dan S, kemudian R, yang pada akhirnya menurunkan P dan B) hanya ditemukan di Bali (Sumatra belum terkonfirmasi). Sedangkan N21a ditemukan di Timor Leste. N21 merupakan garis keturunan Orang Asli (aborigin Melayu: Temuan dan Semelai), yang juga ditemukan di Thailand, Laos, dan Kamboja. N21 dipostulatkan sebagai penanda genetik populasi pertama Sundaland, bersama dengan garis keturunan N yang lain seperti N22, N9 dan R. Sedangkan mtDNA O, S, N13, dan N14 migrasi menuju Sahul, yang akhirnya mendiami bagian utara Australia, namun tidak pernah sampai ke Papua Nugini. Haplogroup N21 lahir tak lama setelah macro-haplogroup N ada di Sundaland sekitar 70-63.000 tahun yang lalu. Meskipun sangat jarang ditemukan di Indonesia, namun keragaman genetiknya lebih tinggi dari garis keturunan aborigin Melayu, yang sebenarnya juga cukup beragam dalam filogenetik N21, menunjukkan bahwa N21 berasal dari kepulauan Nusantara (Sundaland) yang kemudian migrasi ke Semenanjung Melayu, Thailand, Laos dan Kamboja.
N22 (3 mutasi dari macro-haplogroup N) ditemukan di Bali. N22 juga ditemukan pada populasi Temuan, sedikit di Filipina, Sumba dan Sumatra. Di Indonesia mungkin lebih jarang, namun keragamannya lebih tinggi. Haplogroup N21 dan N22, yang terisolasi pada populasi aborigin Melayu merepresentasikan aliran gen dari kepulauan Nusantara (Sundaland).
R21 (4 mutasi dari R, kerabat dekat R22) hanya ditemukan pada populasi Mentawai dan Dayak (Barito) di Kalimantan Selatan. Ditemukan juga pada populasi Negrito Semang (Batek, Jahai, dan Mendriq) dan Temiar Senoi. Bisa jadi bahwa populasi Mentawai tersebut berkerabat dekat dengan Negrito Semang, sama-sama migrasi dari Sundaland. R21 juga kerabat dekat R9 bersama dengan R*, hanya ditemukan pada populasi Sumatra, Mentawai dan Flores (Selain mengalami diversifikasi di Semenanjung Melayu, bukti menunjukkan bahwa R juga mengalami diversifikasi di Wallacea). Selain itu, R9 ditemukan Jahai Semang dan Temiar Senoi. R21 lahir dari leluhurnya, haplogroup R, ~60.000 tahun yang lalu (Macaulay et al., 2005: Single, rapid coastal settlement of Asia revealed by analysis of complete mitochondrial genomes; Jinam et al., 2012: Evolutionary history of continental southeast Asians: ‘Early train’ hypothesis based on genetic analysis of mitochondrial and autosomal DNA data) , meskipun hampir seumuran dengan R9 (2 mutasi dari R), hanya lebih muda. Seperti M21, R21 adalah populasi pribumi Semang/Senoi dan merepresentasikan leluhur yang hidup pada era Pleistocene Akhir di Semenanjung Melayu yang tidak menyebar luas. Ada kemungkinan bahwa mereka hidup terisolasi sejak Pleistocene Akhir. Semang, yang menampilkan phenotype paling konsisten di areanya, memiliki proporsi garis keturunan pribumi yang tinggi: M21a, M21b, dan R21 (Hill et al., 2006; Oppenheimer 2011).
R22 (3 mutasi dari R) ditemukan di Nias, Mentawai, Jawa, Bali, Lombok, Bajo, Sumba, Flores, serta Lembata. Juga ditemukan di daratan Asia Tenggara (Kamboja dan Thailand) dan kepulauan Nicobar (etnis Shompen, yang juga didominasi mtDNA B5a dan R12). R22 paling beragam di kepulauan Nusantara, dan basal haplogroup hanya ditemukan di sekitar Bali, Lombok, Alor dan Sumba (Hill et al., 2007), menunjukkan bahwa hal itu bisa menjadi penanda pribumi untuk Nusa Tenggara. Jika haplogroup N21, R22, M45, M46, M47, dan M21d dan M* tersisa memang mewakili haplogroup pribumi, maka ini menunjukkan bahwa sekitar seperlima dari penduduk Indonesia saat ini dapat ditelusuri nenek moyang mereka kembali ke populasi pertama prasejarah Sundaland.
R23 (3 mutasi dari R) ditemukan di Bali dan Sumba. Di Kamboja juga ditemukan namun bisa jadi migran yang berasal dari Indonesia. R23 adalah garis keturunan R(xR9) di Wallacea (Flores), memisahkan diri dari leluhurnya (R) menuju Sumba dan Bali.
Keragaman basal haplogroup R tertinggi terdeteksi di sekitar Wallacea selatan. Di samping itu, garis keturunan R lebih beragam ditemukan di sekitar Wallacea selatan, seperti R22, R23, R14 dan P. Delapan garis keturunan haplogroup R ditemukan disekitar Wallacea (P, R21’12, R14, R22, R23, R11’B6, B4’5 dan R9/F). Bisa disimpulkan bahwa mtDNA R mengalami diversifikasi di Wallacea (Nusa Tenggara). Jadi tidak heran jika leluhur mtDNA haplogroup B4 (leluhur penutur Austronesia) memang sudah ada di Nusa Tenggara dalam bentuk mtDNA B4.
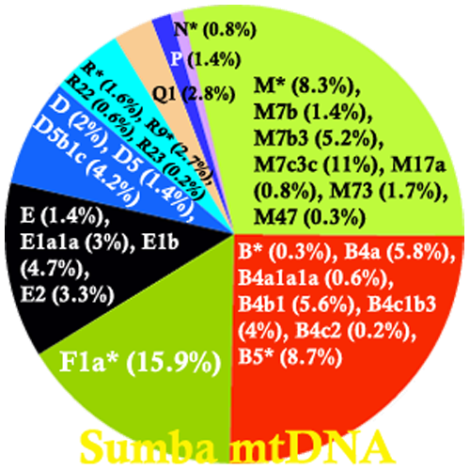
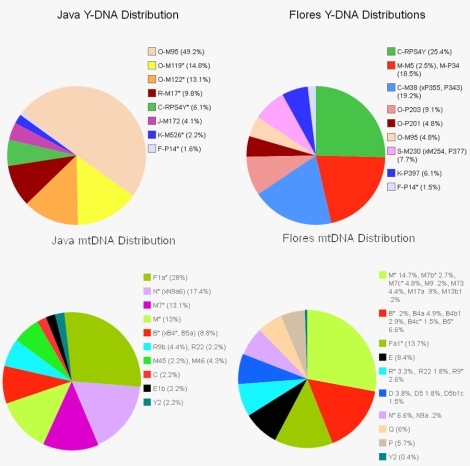
Perhatikan sebaran mtDNA populasi Flores (diagram di bawah), maka akan kita temukan bahwa garis keturunan mtDNA haplogroup R sangat beragam, bahkan pendahulunya (mtDNA haplogroup N) juga masih terdeteksi jejaknya. Dan jika digabungkan dengan sebaran mtDNA di Lembata, Alor, Solor, Pantar, Sumba, dan Timor, maka kita akan menemukan bahwa populasi tertua dari garis keturunan mtDNA M dan N semua masih terdeteksi di Nusa Tenggara. Prasyarat daerah asal-usul mtDNA haplogroup R terpenuhi di Wallacea (Tumonggor et al. (2013) mencatat: mtDNA Flores mencakup 38 haplogroup, Sumba 35 haplogroup dan Timor 31 haplogroup). Segitiga Flores, Sumba dan Timor merupakan lokasi penting di mana terjadi diversifikasi mtDNA haplogroup M dan N yang selanjutnya diversifikasi R. Artinya, populasi tertua di Australasia (Wallacea dan Sahul) dimulai di segitiga Wallacea tersebut.
Basal R12 pada aborigin Australia terpisah dari R21 Negrito Semang, namun keragaman tertinggi terdeteksi di wilayah antara keduanya (Indonesia).
R14 hanya ditemukan di Bali, Bugis, Flores, Lembata, dan Timor Leste. Basal mtDNA haplogroup R di daratan Asia Tenggara tidak memiliki garis keturunan R14.
Haplogroup B tersebar dengan frekuensi tinggi di daratan Asia Tenggara (20,6%), kepulauan Nusantara dan Filipina serta Taiwan (15,5%), Oceania (10,2%), Asia Timur (10,5%) dan Amerika (24%), namun sedikit (0,1-1%) ditemukan di wilayah Volga-Ural, Kaukasus, Asia Barat dan Selatan. Haplogroup B juga terdeteksi sangat kecil di beberapa populasi Eropa, sekitar 3% di Asia Utara dan Tengah, meskipun mencapai >10% di beberapa populasi Siberia.
Haplogroup B diidentifikasi dengan adanya penghapusan 9-bp (base-pair) dalam area intergenik COII/tRNALys mtDNA, secara berulang, dengan transisi pada posisi 16189, menentukan kluster monofiletik secara pasti, yang terdiri dari subhaplogroup B4 dan B5. Dalam penelitian lain, B4’5 bercabang menjadi B4, B5 dan R24. B4 akhirnya menyebar dan keragaman basal haplogroup seharusnya juga berada di sekitar Wallacea. B4 banyak ditemukan di Nias, Jawa, Bali, Flores, Sumba, Sulawesi dan Lembata, juga di Taiwan dan Filipina. B5 banyak dijumpai di Sumatra dan Jawa. is commonest in Nias, Mentawai, Jawa, Bali dan Nusa Tenggara. R24 ditemukan di Filipina (Luzon, Visayas, Mindanao) dengan garis keturunan sangat panjang. Hal ini bisa jadi karena proses isolasi berkepanjangan di wilayah Filipina. Di Indonesia, R24 hanya ditemukan di Maluku Utara (bisa jadi migran dari Filipina). Kerabat dekat B4’B5 yang mengalami mutasi pada posisi 16189 dan polimorfisme tambahan pada posisi np 12950, terdeteksi di Asia Tenggara dan Asia Timur, dinamakan haplogroup R11’B6. Kluster R11’B6 kemudian menurunkan R11, yang tidak mengalami penghapusan 9-bp, dan B6, yang mengalami penghapusan tersebut. R11 banyak terdeteksi di China, sedangkan garis keturunan B6 ditemukan di Asia Timur dan Asia Tenggara.
Penelitian pendahulu menempatkan umur mtDNA haplogroup B4 sekitar 44.000 tahun yang lalu, berasal dari wilayah sekitar Asia Timur atau daratan Asia Tenggara, yang kemudian menyebar sekitar daerah pesisir Vietnam menuju Jepang. Haplogroup B4 bercabang sekitar 35.000 tahun yang lalu menjadi: B4a, B5b’d, B4c (dengan subclade B4b, B2, yang hanya ada di populasi Indian Amerika dan berumur 16.000 tahun. Subclade = subgroup dari sebuah haplogroup). Subclade B4a dan B4a1 juga dipercaya berasal dari daratan Asia Tenggara sekitar 24-20.000 tahun yang lalu, namun B4a1a1a yang ditentukan dengan adanya transisi pada posisi np 16247, dikenal dengan Polynesian motif, adalah subclade terbanyak dalam B4a1a dan mencapai fiksasinya di Polynesia. Berdasarkan analisa mtDNA, PM muncul sekitar >6000 tahun yang lalu di sekitar Kepulauan Bismarck, dan populasi pendahulunya >8000 tahun ditemukan hanya di sekitar Near Oceania.
R9/F hanya dua mutasi dari haplogroup R. R9 ditemukan di Sumatra, Mentawai, Flores, dan Sulawesi. Juga di China Selatan serta aborigin Melayu. Haplogroup R9c (satu mutasi dari R9 pada posisi np 16157) ditemukan dengan frekuensi sangat rendah di Asia Timur (<0,5% di China), daratan Asia Tenggara (<1%), Taiwan (1,8%) dan kepulauan Asia Tenggara (<3%), namun meningkat frekuensinya di Filipina (3,3-5,7%) dan di Alor (11,1%). Semua HVS1 R9c1 memiliki mutasi pada posisi np 16157. Berdasarkan analisa mtDNA, garis keturunan dengan mutasi tersebut dikelompokkan dalam R9c1a1, subclade dari R9c1a. Subclade R9c1a tertua berasal dari Asia Timur sekitar 30-37.000 tahun yang lalu, dan subclade R9c1a1 adalah mereka yang berekspansi ke Asia Tenggara sekitar 9000 tahun yang lalu, mirip dengan haplogroup B4c2 dan E1a2. Jika dua haplogroup terakhir terkait dengan penutur proto-Austronesia/proto-Malayo-Polynesia, maka bisa disimpulkan bahwa R9c1a1 adalah populasi yang juga hidup di masa yang sama dengan penutur proto-Austronesia di Alor. Seperti diagram di bawah, bahwa garis keturunan B4, B5, R9c dan kehadiran garis keturunan F1a yang beragam, membuat Wallacea sebagai kandidat kuat lokasi diversifikasi mtDNA haplogroup R, dan kemudian menurunkan hampir semuasubclade-nya: R22, R23, R14, B, P, R9/F di Australasia (minus R21). Dengan demikian, populasi dengan garis keturunan maternal haplogroup R bisa disimpulkan adalah penduduk asli (autochthonous) wilayah Wallacea.
Haplogroup F terkonsentrasi di Taiwan dan Sumatra, F1 di Filipina, Maluku, Nusa Tenggara, Borneo, Jawa, Nicobar (proporsi kecil di India, Sumatra, Malaysia, Vietnam dan Thailand). Jika F1 sudah ada di Indonesia sejak awal, mungkin perlu ditelusuri lebih lanjut asal-usul mtDNA F1a1 (penanda genetik penutur Austroasiatik yang disimpulkan berasal dari wilayah sekitar Laos)
Berkenaan dengan hipotesis tentang asal-usul penutur Austronesia dari Taiwan, maka bagaimana kekerabatan genetik antara Indonesia, Filipina dan Taiwan akan menjadi sangat menarik. Ketiganya memiliki empat haplogroup yang sama (E1a1a, M7b3, M7c3c, dan Y2), yang awalnya dianggap sebagai populasi yang migrasi dari Taiwan sekitar pertengahan Holocene. Anggapan yang salah karena mtDNA E, berasal dari Borneo timur laut / Sulawesi barat laut, migrasi menuju Taiwan dan Filipina pada awal Holocene, dan E1a1a adalah penutur proto-Malayo-Polynesian (berdasarkan genetik populasi kepulauan Mariana di Pasifik yang migrasi sekitar 3500 tahun yang lalu). Garis keturunan Taiwan dan Filipina lebih mirip dengan Indonesia bagian timur daripada bagian barat. Dengan pengecualian Y2 dan M7c3c, yang frekuensinya cukup tinggi di Nias dan Mentawai, bisa jadi akibat pergeseran genetik atau founder effects (migran pertama di daerah tersebut) di wilayah dengan geografi yang kecil dan terisolasi.
Dari keempat haplogroup (E1a1a, M7b3, M7c3c, dan Y2), setidaknya populasi Indonesia dan Taiwan sharing masing-masing satu haplotype, dan haplotypetersebut sama-sama dalam posisi ancestral (yang satu bukan keturunan/leluhur yang lain, melainkan keduanya saudara yang terpisah sejak awal). Dari analisa keempat haplogroup tersebut, arah migrasi dari Taiwan ke Filipina dan Indonesia tidak bisa ditentukan dengan pasti, karena arah yang berlawanan juga sesuai dengan model analisa. Dan lagi, berdasarkan umurnya, adalah tidak mungkin sebaran berasal dari Taiwan, karena populasi pendahulu lebih dulu ada di kepulauan Nusantara. Namun, dari Y-chromosome, leluhur populasi Austronesia di Taiwan dan Indonesia terpisah ketika mereka masih di sekitar Teluk Tonkin. Yang paling mungkin adalah, bahwa terjadi sebaran populasi di kepulauan Nusantara selama era Holocene.
Polynesian motif (PM) yang sering diasosiasikan dengan sebaran Austronesia dari Taiwan, namun sebaran geografisnya janggal. Populasi pendahulu (leluhur) tersebar luas di daratan Asia Tenggara dan kepulauan Nusantara. Sedangkan PM hanya ditemukan rendah di Filipina (0.5%) sedangkan di Indonesia bagian timur (2,3%), meskipun cukup tinggi di Timor (7,4%), dan tidak ditemukan di sebelah barat Bali. Hal tersebut tidak serta merta menjadikan asal-usul PM dari Bismarck Archipelago kemudian migrasi ke arah Indonesia timur (Soares et al. 2011:Ancient voyaging and Polynesian origins), karena tingginya frekuensi PM di Madagascar yang berasal dari garis keturunan PM dari Borneo dan sekitarnya (Kusuma et al. 2015), menjadikan daerah asal-usul PM masih di sekitar Wallacea (Halmahera).
Analisa mtDNA juga bisa mengungkap jumlah populasi. Berdasarkan filogeni garis keturunan mtDNA populasi Asia Tenggara, sinyal pertumbuhan tertinggi terpusat di Wallacea (Bali, Sulawesi, Sumba dan Flores), melemah di ujung timur dan barat kepulauan Nusantara. Pengecualian pada populasi Nias dan Mentawai yang secara statistik jumlah populasi konstan konsisten dengan profil mtDNA. Dua kelompok etnis tersebut juga memiliki keragaman haplotype terendah dibandingkan populasi Asia Tenggara (43,5% wanita Nias mewarisi mtDNA Y2). Pola tersebut akibat pergeseran genetik, sebagaimana populasi Nias dan Mentawai tergolong kecil dibandingkan populasi etnis Indonesia yang lain. Atau, bisa juga akibat dari founder effects (migran pertama di Nias dan Mentawai, kemudian mengisolasi diri dari luar). Para penduduk Nias masih melestarikan tradisi leluhur mereka seperti bangunan megalitik, dan hombo batu (lompat batu). Tradisi bangunan megalitik banyak diasosiasikan dengan budaya Austronesia yang dulu sangat lazim di Indonesia, namun sekarang hanya terbatas pada populasi pinggiran seperti Nias, Mentawai dan Sumba.
Analisa lebih lanjut, dengan mengukur jarak genetik antar populasi, didapat bahwa populasi Indonesia bagian barat terpisah dari Indonesia bagian timur. Jika dibandingkan dengan wilayah sekitar, populasi Indonesia lebih dekat kekerabatan genetiknya dengan daratan Asia Tenggara dan Filipina serta Taiwan, daripada dengan Oceania. Indonesia bagian barat berbeda dengan bagian timur, karena rendahnya mtDNA haplogroup P dan Q (Forster et al., 2001: Phylogenetic star contraction applied to Asian and Papuan mtDNA evolution). Apakah garis keturunan mtDNA P dan Q tersebut keturunan pribumi Sundaland yang tidak meninggalkan tanah leluhurnya, atau migran dari Papua di kemudian hari, masih tidak jelas. Di antara populasi Asia, populasi Indonesia terkelompok lebih dekat dengan populasi Filipina dan Vietnam (bisa jadi termasuk Laos dan Kamboja), dan lebih jauh dari Taiwan.
Dengan membandingkan penanda genetik paternal dan maternal, disimpulkan oleh Tumonggor et al. (2013) bahwa wanita Sundaland menyebar lebih luas di masa lalu dibandingkan dengan pria. Salah satu kemungkinan adalah sistem sosial patrilokal, di mana para pria menetap di tanah kelahirannya, sementara wanita berpindah mengikuti suami. Menariknya, populasi penutur Austronesia menganut sistem sistem matrilokal. Pola mtDNA/Y-DNA tersebut bisa jadi akibat pergeseran budaya, dan karena itu hanya berlangsung beberapa ribu tahun saja. Alternatifnya, patrilokal menjadi standar sistem kemasyarakatan jangka panjang, dengan transisi matrilokal sementara selama ekspansi Austronesia. Perbedaan laju mutasi sudah pasti akan membingungkan analisa tersebut; STR memiliki laju mutasi yang tinggi dan seharusnya hanya digunakan dalam jangka waktu pendek, bahkan jika dibandingkan dengan mtDNA yang mutasinya dipercepat.
Sejarah populasi Indonesia secara umum terbagi atas lima gelombang migrasi berdasarkan Y-chromosome (Y-DNA), namun mtDNA bisa juga mengikuti model tersebut, berdasarkan proporsi masing-masing haplogroup dalam kelompok etnis yang ada.
Gelombang pertama, menurut Karafet et al., (2010: Major east-west division underlies Y-chromosome stratification across Indonesia) berdasarkan garis keturunan maternal diwakili oleh mtDNA haplogroup M17a, M73, M47, N21, N22, R21, R22, dan R23, yang bisa ditelusuri kembali ke populasi macro-haplogroup M dan N serta R yang ada di Asia Tenggara.
Gelombang kedua, menurut Karafet et al., diwakili garis keturunan B4a, B4b, B4c, B4c1b3, B5a, B5b, B5b1, D dan E) yang berumur ~40.000 tahun. Bandingkan dengan Gelombang kedua dari Sunda ke Sahul (garis keturunan macrohaplogroup N yang menuju Australia, garis keturunan mtDNA R: P, dan B di Wallacea dan Sahul; garis keturunan mtDNA M: M29’Q, M27, M28, serta M42a, M14 dan M15 di Australia). Berdasarkan keragaman yang berbeda dari masing-masing garis keturunan tersebut, sangat tidak mungkin bahwa setiap peristiwa migrasi membawa masing-masing garis keturunan tersebut ke Indonesia. Garis-garis keturunan maternal tersebut memang sangat terkait dengan populasi Orang Asli di Semenanjung Melayu, namun berdasarkan data genetik, garis keturunan tersebut juga ditemukan di Indonesia. Mereka adalah populasi pertama Sundaland yang menyusuri sungai-sungai purba yang menghubungkan Semenanjung Melayu dengan Sundaland.
Gelombang ketiga, menurut Karafet et al. adalah migrasi Neolitik ke dan di sekitar Asia Tenggara. Beberapa diantaranya bisa jadi termasuk migrasi dari Taiwan, di bagian lain adalah perpindahan populasi dari pulau satu ke pulau yang lain di kepulauan Nusantara. Gelombang ketiga diwakili oleh garis keturunan mtDNA haplogroup M7b3, E1a1a, M7c3c, dan Y2. Saya berpendapat, bahwa gelombang ketiga di wakili oleh mtDNA haplogroup E (penanda genetik proto-Austronesia/proto-Malayo-Polynesia sebelum era Holocene) dan B4a1a yang dianggap penutur proto-Austronesia, bersama dengan mtDNA F1a1a dan N9a6 yang merupakan penanda genetik penutur Austroasiatik. Hal ini diperkuat oleh sebaran Y-DNA O2a1-M95, yang merupakan penanda genetik Austroasiatik. Gelombang ketiga ini seharusnya terjadi di masa LGM (~21-11.500 tahun yang lalu, sesuai dengan umur mtDNA dan Y-DNA yang terlibat).
Gelombang keempat, menurut Karafet et al. adalah migrasi di jaman sejarah, termasuk mereka yang terlibat dalam perdagangan maritim masa lalu, seperti India, Arab dan China. Meskipun frekuensinya rendah, garis keturunan paternal yang mewakili gelombang keempat ini banyak ditemukan di Indonesia bagian barat, sesuai dengan sebaran agama Hindu oleh para brahmana dari India timur, dan mungkin keturunan raja-raja India yang mendirikan dinasti di kerajaan-kerajaan Hindu di Nusantara, seperti dinasti Warman dari India selatan. Y-DNA H dan R1a1 dari India, dan J-M172 dari Arab, serta O-M134 dari China. Menariknya, tidak ditemukan sebaran mtDNA dari masa sejarah dari luar Indonesia. Hubungan dagang dan penyebaran agama Hindu di Nusantara tidak melibatkan wanita migran. Konsekuensinya, pria migran tersebut mengambil istri dari populasi lokal. Saya berpandangan bahwa gelombang ke empat versi Karafet et al. adalah gelombang kelima, sedangkan gelombang ketiga versi Karafet adalah gelombang keempat. Karafet et al. tidak mempertimbangkan sebaran garis keturunan penutur Austroasiatik, yang frekuensinya cukup tinggi di Indonesia bagian barat (50-60%).
Catatan menarik dari Karafet et al. adalah bahwa garis keturunan mtDNA yang ditemukan di hampir seluruh wilayah Indonesia adalah M7c3c (yang menurutnya adalah penanda genetik penutur Austroasiatik), garis keturunan yang juga ditemukan di Taiwan dan Filipina, namun tidak ditemukan di Oceania (Pasifik). Populasi Austronesia lebih terkarakterisasi dari keragaman genetiknya dibanding dengan kesamaan garis keturunan secara genetik. Dengan temuan ini, penutur Austronesia tidak hanya diwakili oleh PM (B4a1a1a), sehingga bisa jadi B4a1a1a menyebarkan proto-Austronesia, sedangkan M7c3c lebih terkait dengan Malayo-Polynesia.
Sumber:
https://motherlanders.wordpress.com/2015/05/04/jalur-lintas-genetik-purba-antara-asia-dan-pasifik/
No comments:
Post a Comment